SBOL: Open-Source-basierter Austausch für Biotech-Enthusiasten
Gerade in der aktuellen Zeit erleben wir, wie wichtig der Austausch zwischen Wissenschaftlern ist. Nahezu täglich gibt es neue Erkenntnisse, die untereinander besprochen und diskutiert werden müssen.
Dies gilt für die Lage in der aktuellen Pandemie genauso wie allgemein betrachtet in der (biotechnischen) Forschung.
Ein Beispiel: Der aktuelle Stand der Forschung macht es möglich, dass nicht nur das Genom von Bakterien und Viren vollständig kartografiert – bzw. wie es in der Fachsprache heißt „sequenziert“ – werden kann, sondern auch von Menschen und Tieren. Es geht noch weiter: Gene können nicht nur sequenziert, sondern auch vollständig synthetisiert und editiert werden. Die meisten kennen sicher die „Big Bang Theory“-Folge, in der ein leuchtender Goldfisch gezeigt wird. In dieser Darstellung ist das wissenschaftlich nicht korrekt, aber es ist möglich: Die Fische leuchten zwar nicht selbst, aber fluoreszieren, weil man ihnen das Gen einer fluoreszierenden Qualle oder Seeanemone eingesetzt hat. Durch dieses Wissen haben Anbieter wie PetCo & Co. so einen „GloFish“ mittlerweile in ihrem Angebotssortiment.
Um sich beispielsweise über solche Erkenntnisse mit anderen Biotechnologie Enthusiasten wie diese besser und unkompliziert austauschen zu können, hat sich eine Gruppe von Wissenschaftlern zusammengeschlossen und einen internationalen Standard entwickelt: Die Synthetic Biology Open Language, kurz SBOL. SBOL teilt sich hintergründig in zwei Teile:
Auf der einen Seite ist der grafische Teil „SBOL Visual“: Dieser beschreibt, welche Symbole für welche funktionalen Komponenten der DNA, RNA usw. – kurz dem „Zentralen Dogma der Molekularbiologie“ plus X – benutzt werden. Auf der anderen Seite ist der datenverarbeitende Teil SBOL Data. Dieser nutzt bereits existierende Prinzipien aus der Informationstechnologie wie z.B. die automatische Umwandlung von Basenpaarsequenzen in Symbole.
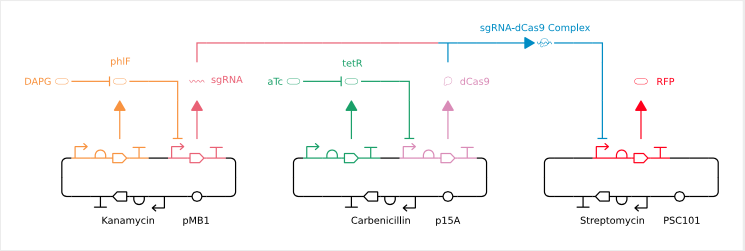
Bild Beschreibung: Drei DNA Ringe – auch Plasmide oder Mini-Chromosomen genannt, darauf verschiedene Gene und die Wechselwirkungen zwischen ihnen und ihrer Produkte: Jeweils ein geknickter Pfeil(Promotor), ein Halbkreis(Ribosom-Bindestelle), ein voller Pfeil(Kodierender Teil) und ein T(Terminator) jeweils in den Plasmiden ergeben ein Gen. Die schwarzen Bereiche der Plasmide sind zuständig für organisative Aufgaben wie Selbsterhalt und Reproduktion der Plasmide im Lebewesen.
Vom rot markierten Gen aus wird ein rot fluoreszierendes Protein erzeugt – RFP. Falls sich das Protein dCas9(rosa) mit einer sgRNA(koralle) verbindet(blau), wird die Produktion von RFP blockiert weil die Verbindung den Promoter unzugänglich macht. Vom grün und orange markierten Gen aus werden Proteine(phlF und tetR) gebildet die wiederum entsprechend die Synthese von dCas9 und der sgRNA verhindern. Mit der Zugabe von DAPG(orange) oder aTc(grün) kann man diese Proteine so manipulieren, dass sie die Produktion von sgRNA und dCas9 nicht mehr unterbinden, was letztendlich einen chemischen An-Aus-Schalter darstellt.
Aber was nutzt schon eine „offene Sprache“, wenn sie nicht einfach zugänglich ist. Kaum ein Biologe, Mikrobiologe oder Genetiker „spricht“ Python, kann also beispielsweise nicht ohne Weiteres mit der Bibliothek DNAplotlib arbeiten. Es musste also was Einfacheres her. Und es wurde etwas Einfacheres geliefert: Mittlerweile ist man als Forscher nicht mehr darauf angewiesen eine Programmiersprache zu lernen, um die Symbole professionell nutzen zu können. Denn dem SBOL Paket wurde eine offizielle Schriftart hinzugefügt.
Nach wie vor wird weiter an SBOL gearbeitet und aufgrund ständig neuer wissenschaftlicher Erkenntnisse stetig neue Symbole und Funktionalitäten hinzugefügt. Zudem ist geplant, die Symbole in die Unicode Tabelle zu übernehmen.
Wer aktiv mitentwickeln möchte, kann dies im offiziellen GitHub Projekt dazu tun. Weitere Infos dazu gibt es auch auf der offiziellen SBOL-Seite oder in diesem Paper.
Der ATIX-Crew besteht aus Leuten, die in unterschiedlichen Bereichen tätig sind: Consulting, Development/Engineering, Support, Vertrieb und Marketing.